Commits on Source (14)
-
Ondřej Novák authored
-
Ondřej Novák authored
-
Ondřej Novák authored
-
Ondřej Novák authored
-
Ondřej Novák authored
-
Ondřej Novák authored
-
Ondřej Novák authored
-
Ondřej Novák authored
-
Ondřej Novák authored
-
Ondřej Novák authored
-
Ondřej Novák authored
-
Ondřej Novák authored
-
Ondřej Novák authored
-
Ondřej Novák authored
Showing
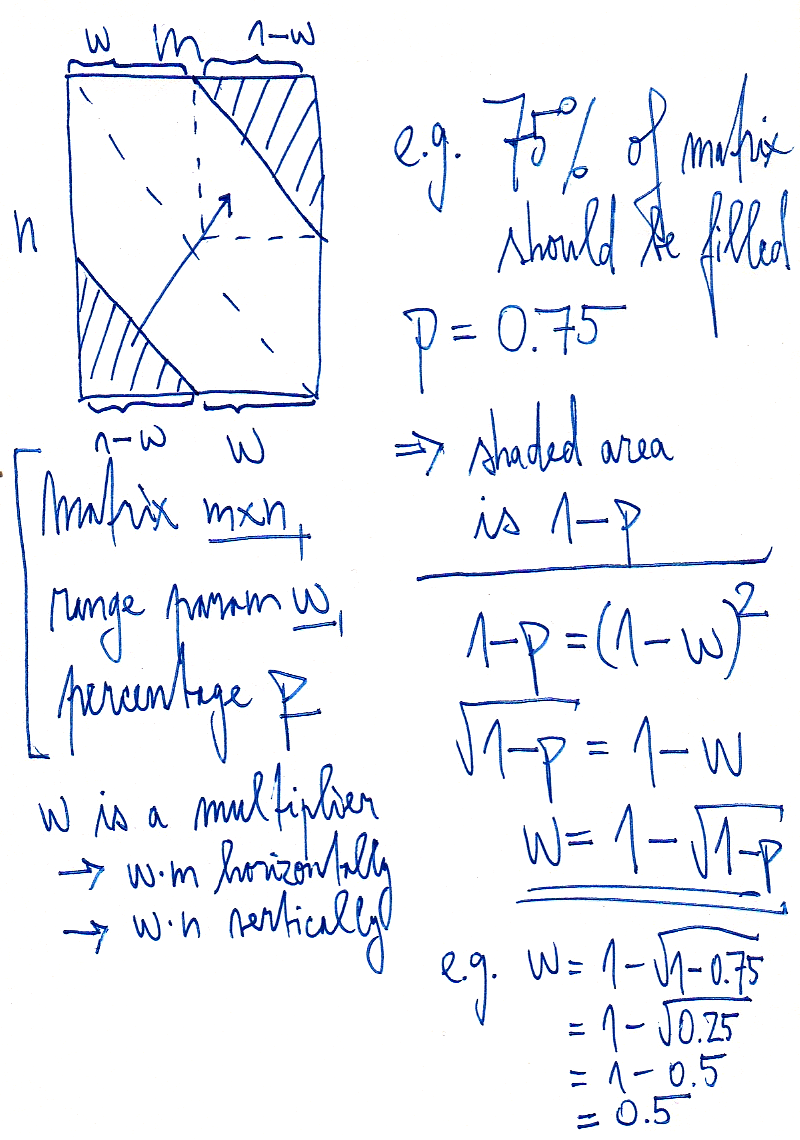
- Makefile 2 additions, 1 deletionMakefile
- docs.md 76 additions, 0 deletionsdocs.md
- docs_w.png 0 additions, 0 deletionsdocs_w.png
- dtw.c 42 additions, 20 deletionsdtw.c
- readme.md 5 additions, 9 deletionsreadme.md
- test.txt 2740 additions, 2740 deletionstest.txt
- time.sh 1 addition, 1 deletiontime.sh
- time/time.txt 32 additions, 30 deletionstime/time.txt
- time/time005.txt 99 additions, 9 deletionstime/time005.txt
- time/time010.txt 92 additions, 2 deletionstime/time010.txt
- time/time025.txt 90 additions, 0 deletionstime/time025.txt
- time/time050.txt 90 additions, 0 deletionstime/time050.txt
- time/time100.txt 90 additions, 0 deletionstime/time100.txt
- time/time_old.txt 0 additions, 30 deletionstime/time_old.txt
- time/time_opt.txt 0 additions, 30 deletionstime/time_opt.txt
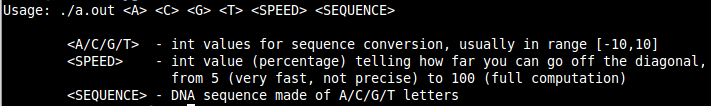
- usage.png 0 additions, 0 deletionsusage.png
- usage.txt 8 additions, 0 deletionsusage.txt
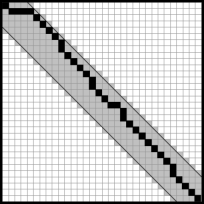
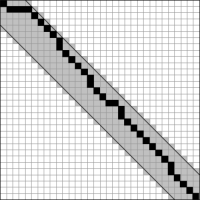
- web/img/matrix.png 0 additions, 0 deletionsweb/img/matrix.png
- web/img/plot.png 0 additions, 0 deletionsweb/img/plot.png
- web/img/results.png 0 additions, 0 deletionsweb/img/results.png
docs.md
0 → 100644
docs_w.png
0 → 100644
373 KiB
This diff is collapsed.
time/time_old.txt
deleted
100644 → 0
time/time_opt.txt
deleted
100644 → 0
usage.png
deleted
100644 → 0
21 KiB
usage.txt
0 → 100644
| W: | H:
| W: | H:


| W: | H:
| W: | H:


web/img/results.png
0 → 100644
167 KiB